La révolution du très haut débit
Le développement depuis 2009 des technologies de séquençage de nouvelle génération (NGS, Next Generation Sequencing) constitue une révolution technologique sans précédent.
Alors que le séquençage d’un seul génome humain a nécessité 10 ans de collaborations internationales (1993-2003) et 2,7 milliards de dollars, il est désormais possible de séquencer l'exome humain en l’espace de quelques semaines, voire de quelques jours, pour un coût inférieur à 1 000 euros. Le séquençage de génomes humains entiers, composé de 3 milliards de paires de bases, a déjà été réalisé à visée médicale par quelques équipes aux États-Unis, au Royaume-Uni, aux Pays-Bas et en France.
Grâce notamment au multiplexage (séquençage de plusieurs échantillons, identifiés par code-barre, au sein d'un même run), il est possible d'augmenter les capacités de séquençage et d'en diminuer le coût. Ces avantages sont essentiels à la mise en place d'un diagnostic génétique étendu au plus grand nombre de patients.
Parallèlement, les progrès en matière de techniques de gestion et d'analyse des données massives (Big Data) et de nouvelles infrastructures de stockage doivent permettre d'absorber cette augmentation des capacités de séquençage afin de prendre en charge des données de plus en plus nombreuses.
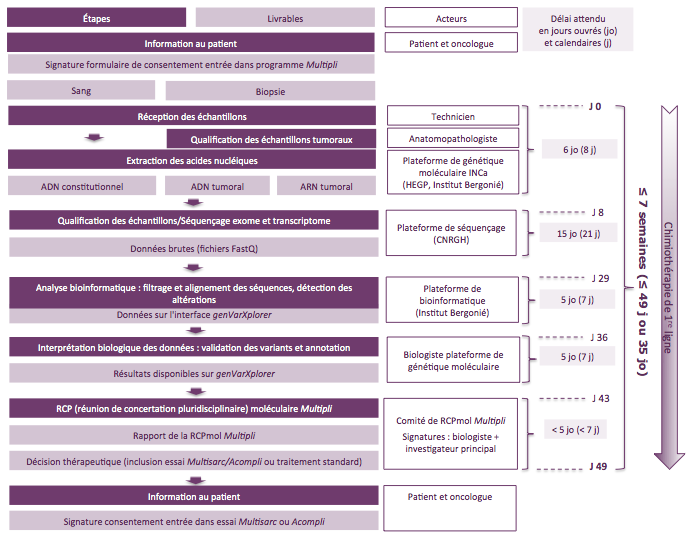
Le dispositif MULTIPLI
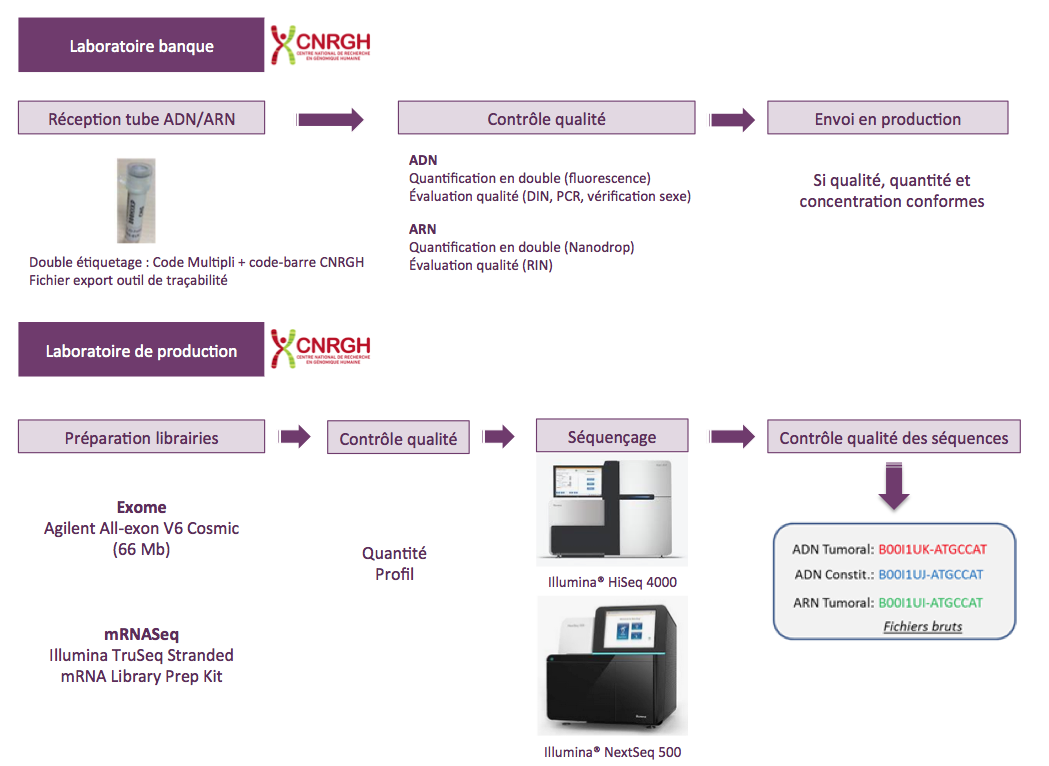
Parcours génomique des échantillons
Procédure de séquençage
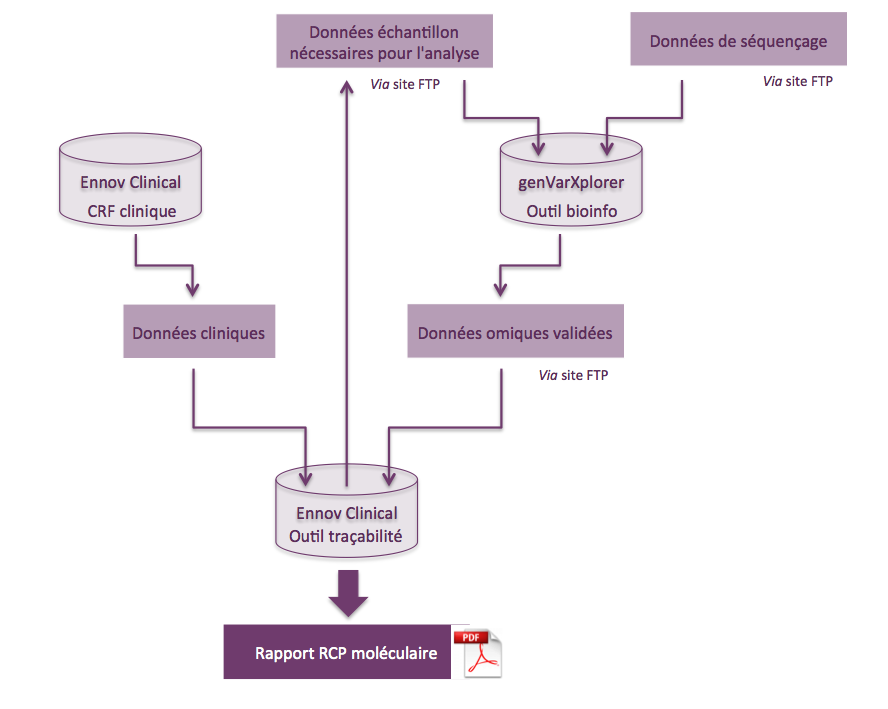
Circulation des données
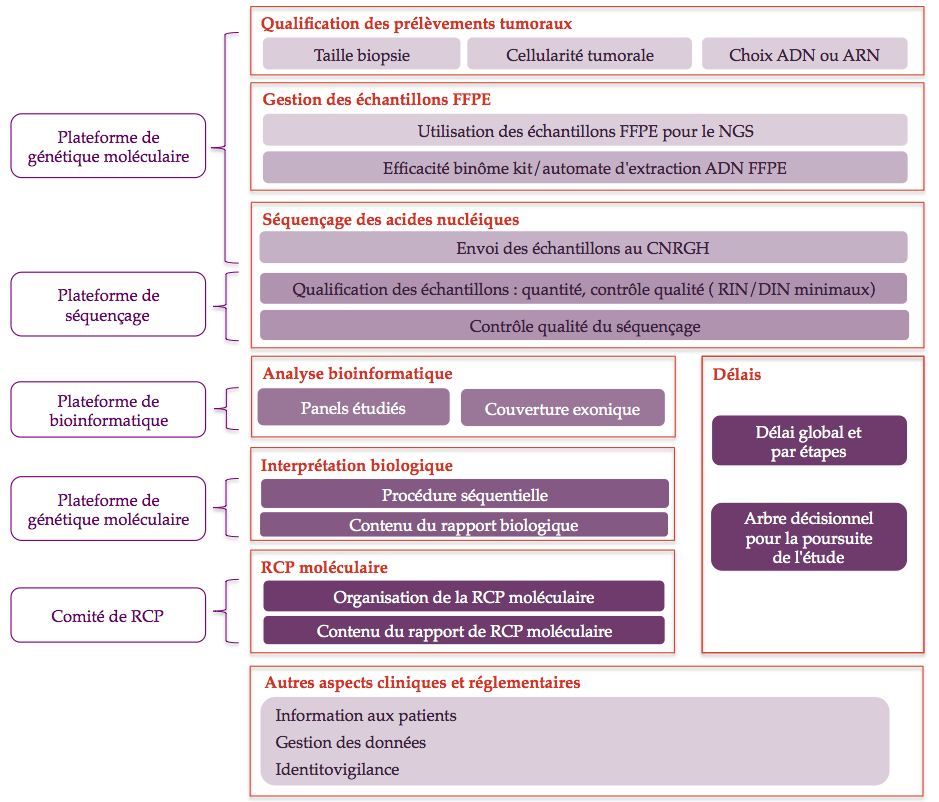
Premiers verrous identifiés et levés
Des travaux préliminaires ont permis au comité de pilotage de MULTIPLI de rédiger les procédures nécessaires au bon déroulement de l'analyse génomique à haut débit. Une évaluation en situation réelle de la faisabilité du séquençage à haut débit a été menée afin d'évaluer la robustesse des procédures. A l’issue de cette évaluation, 30 verrous ont pu être identifiés et levés : ils constituent autant de recommandations pour s'assurer du bon déroulement de la phase de séquençage dans le programme MULTIPLI.